
Un trabajo recientemente publicado en la revista Journal of Antimicrobial Chemotherapy describe los elementos genéticos móviles que diseminan la resistencia a antibióticos macrólidos en Streptococcus pyogenes. Este trabajo liderado por Carmen Ardanuy, investigadora del CIBERES en el Hospital Universitario de Bellvitge, explora la adquisición de resistencia a macrólidos en los últimos 21 años; un periodo en el que se observa un claro aumento en resistencia debido, principalmente, a la presencia de elementos genéticos móviles.
S. pyogenes se asocia principalmente con la faringoamigdalitis bacteriana pero también es conocida como la bacteria “come carne” responsable de fascitis necrotizante. Aunque los beta-lactámicos siguen siendo el tratamiento de elección para este tipo de infecciones, en pacientes alérgicos a la penicilina, los macrólidos son la principal alternativa, por lo que la vigilancia de las tasas de resistencia es importante para mantener la eficacia clínica.
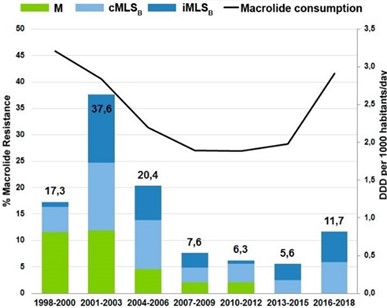
En este estudio se observa cómo, después de casi una década con tasas de resistencia a macrólidos inferiores al 8% y en descenso, se ha producido entre los años 2016 y 2018 un aumento hasta el 11.4% en el nivel de resistencia. Este claro repunte se ha asociado con la dinámica clonal de unos linajes portadores de elementos de resistencia a los macrólidos, pero también se ha visto afectado por altos niveles de consumo de antibióticos que
pueden favorecer la aparición y propagación de organismos resistentes.
La caracterización de estas cepas resistentes mediante Whole Genome Sequencing ha permitido estudiar el entorno genético que moviliza los genes de resistencia y describir tres estructuras móviles nuevas, estando una de ellas directamente asociada con un fenotipo de multiresistencia. Entre las estructuras nuevas encontradas, hay una que es capaz de albergar diferentes genes de resistencia a macrólidos, tetraciclinas, cloranfenicol y cotrimoxazol dando lugar a un fenotipo multirresistente que preocupa.

Este trabajo ha sido posible gracias a la colección histórica de S. pyogenes del Servicio de Microbiología del Hospital Universitari de Bellvitge y a la experiencia del equipo en análisis de Whole Genome Sequencing. El estudio se ha llevado a cabo gracias a las ayudas del Fondo de Investigación en Salud y el CIBER de Enfermedades Respiratorias.
Referencia: doi: 10.1093/jac/dkab130.
Dàmaris Berbel, Jordi Càmara, Aida González-Díaz, Meritxell Cubero, Guillem López de Egea, Sara Martí, Fe Tubau, M Angeles Domínguez, Carmen Ardanuy. Deciphering mobile genetic elements disseminating macrolide resistance in Streptococcus pyogenes over a 21 year period in Barcelona, Spain. J Antimicrob Chemother. 2021; 76(8):1991-2003.